

|
基因芯片技术服务 DNA甲基化芯片 DNA羟甲基化芯片 ChIP-chip |
NGS测序技术服务 DNA甲基化测序 DNA羟甲基化测序 染色质免疫共沉淀测序 |
PCR技术服务 MeDIP-qPCR hMeDIP-qPCR ChIP-qPCR |
| TMT标记定量技术 非标定量技术 |
蛋白修饰 TMT标记定量磷酸化 非标定量磷酸化 |
RNA/蛋白-蛋白相互作用 RNA-蛋白相互作用 蛋白-蛋白相互作用 |
相关产品
rtStar™ First-Strand cDNA Synthesis Products Arraystar SYBR® Green Real-time qPCR Master Mix相关服务
染色质免疫共沉淀测序 DNA甲基化测序 DNA羟甲基化测序 RNA m6A甲基化测序 ChIP-chip DNA甲基化芯片 DNA羟甲基化芯片 ChIP-qPCR MeDIP-qPCR hMeDIP-qPCR RNA测序相关资源
表观遗传学与疾病 表观遗传调控与肿瘤形成的分子机制 DNA甲基化与癌症研究 Cell子刊:Arraystar DNA甲基化芯片用于干细胞移植改善骨质疏松表观机制的研究 5羟甲基化的研究背景及与疾病的关系
| 芯片 | 规格 | 描述 |
|---|---|---|
| NuRNA™ Human Epitranscriptomics PCR Array | 384-well(4*96)/ plate | 同时检测89个已知的或潜在的表观转录修饰相关蛋白 |
近年来,RNA修饰所介导的基因表达调控不断取得突破性进展,直接导致了表观转录组学(Epitranscriptomics)的诞生。最新研究发现,m6A、m5C、m1A、hm5C、甲尿嘧啶(ψ)与肌苷(I)等多种表观转录修饰存在于mRNA上,影响mRNA的代谢与功能[1],是一种重要的基因表达调控方式(图1)。这些表观转录组修饰由特定的酶添加(Writer)或去除(Eraser),可被特定的蛋白识别(Reader)。表观转录修饰水平的异常,以及相关酶或蛋白的异常,与许多疾病的发生紧密关联。
m6A是真核生物mRNA上含量最丰富的表观转录修饰,参与调控mRNA的二级结构、剪切、成熟、细胞定位与翻译等过程。m6A由甲基转移酶复合物(包含METTL3,METTL14,WTAP,KIAA1429,RBM15,RBM15B)催化产生,可被去甲基化酶ALKBH5或FTO去除。目前已发现了多种特异性识别m6A位点的蛋白或复合物,包括YTH家族蛋白(YTHDF1-3,YTHDC1)、转录起始复合物eIF3、核糖核蛋白(HNRNPA2B1,HNRNPC)以及RNA结合蛋白SRSF2(表1)。m6A的动态调控在减数分裂与细胞多潜能分化等过程中发挥着重要作用[3]。与正常组织相比,肿瘤干细胞中的m6A修饰水平显著升高。高水平的m6A增强了多个关键肿瘤基因的蛋白表达,从而促进肿瘤干细胞的增殖与肿瘤的发生发展,且常与不良预后有关。此外,METTL3[4]、FTO[2]及ALKBH5[5]等表观转录相关蛋白因子在肿瘤的发生发展中也发挥着关键性作用。在病毒RNA上也检测到了m6A修饰,其与病毒感染及复制有关。
显而易见,表观转录修饰与表观转录修饰相关蛋白在基因表达调控中发挥着至关重要的作用。然而,目前对其研究才刚刚起步。为了帮助研究人员更方便快捷的进行表观转录组学研究,Arraystar开发了市场上首款检测表观转录修饰相关蛋白因子的PCR芯片。此款芯片可同时检测89个已知的或潜在的表观转录修饰相关蛋白,是表观转录研究的强有力工具。
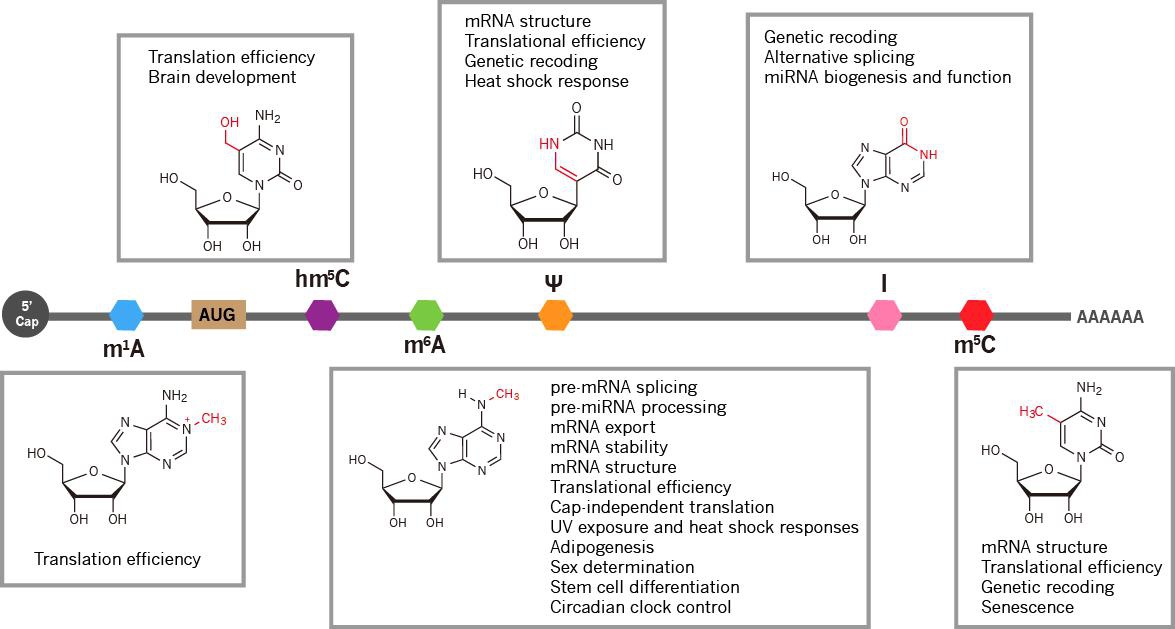
图1.mRNA表观转录修饰及其生物学功能。
| m6A | m5C | I | ψ | m1A | ||
|---|---|---|---|---|---|---|
| Writers | KIAA1429, METTL3, METTL4, METTL14, RBM15, RBM15B, WTAP | NSUN2, TRDMT1 | ADAR, ADARB1, ADARB2 | DKC1, PUS1, PUS3, PUS7, TRUB1 | None identified | |
| rasers | ALKBH5, FTO | TET1, TET2, TET3 | None identified | None identified | ALKBH1, ALKBH3 | |
| Readers | YTH Family, eIF3 complex, HNRNPA2B1, HNRNPC, SRSF2 | None identified | None identified | None identified | None identified |
表1. 已发现的人属mRNA修饰酶(writer)、去修饰酶(Eraser)和修饰识别蛋白(Reader)。
参考文献
1. Gilbert,
W.V., et al. (2016). Messenger RNA modifications: Form, distribution, and
function. Science 352, 1408-1412, PMID:27313037.
2. Iles, M.M., et al. (2013). A variant in FTO shows association with
melanoma risk not due to BMI. Nature genetics 45, 428-432, 432e421,
PMID:23455637.
3. Klungland, A., et al. (2016). Reversible RNA modifications in meiosis
and pluripotency. Nature methods 14, 18-22, PMID:28032624.
4. Lin, S., et al. (2016). The m(6)A Methyltransferase METTL3 Promotes
Translation in Human Cancer Cells. Molecular cell 62, 335-345, PMID:27117702.
5. Zhang, C., et al. (2016). Hypoxia induces the breast cancer stem cell phenotype
by HIF-dependent and ALKBH5-mediated m(6)A-demethylation of NANOG mRNA.
Proceedings of the National Academy of Sciences of the United States of America
113, E2047-2056, PMID:27001847.
•覆盖度广—覆盖了所有目前已知的表观转录修饰相关蛋白
•转录本水平检测—检测编码Uniprot经典蛋白的转录本
•严谨性强—所有引物都通过了多样本严格验证
•快速简易—即用型384孔板规格,只需将cDNA与qPCR
Master Mix混合试剂加入PCR板中,4小时内得到实验结果。cDNA无需预先扩增。
|
表观转录修饰相关蛋白检测列表 |
|
m6A Writers: KIAA1429, METTL14, METTL3, METTL4, RBM15, RBM15B, WTAP Readers: DGCR8, EIF3A, EIF3B, ELAVL1, HNRNPA2, HNRNPB1, HNRNPC1, HNRNPC2, SRSF2, YTHDC1, YTHDC2, YTHDF1, YTHDF2, YTHDF3 Erasers: ALKBH5, FTO m1A Writers: HSD17B10, KIAA0391, TRMT10C, TRMT6, TRMT61A, TRMT61B Erasers: ALKBH1, ALKBH3 m5C Writers: DNMT1 , DNMT3A, DNMT3B, DNMT3L, NOP2, NSUN2, NSUN3, NSUN4, NSUN5, TRDMT1 Readers: MECP2, UHRF1 Erasers: TET1, TET2, TET3 hm5C Writers: TET1, TET2, TET3 Readers: CHTOP, EGR1, ERH, HMCES, MEP50, MGME1, NEIL1, PRMT1, PRMT5, SMUG1, TDG, THYN1, WDR76, WT1 Inosine Writers: ADAD1, ADAD2, ADAR, ADARB1, ADARB2, ADAT1, ADAT2, ADAT3 Pseudouridine Writers: DKC1, GAR1, NAF1, NHP2, NOP10, PUS1, PUS10, PUS3, PUS7, PUS7L, PUSL1, RPUSD1, RPUSD2, RPUSD3, RPUSD4, TRUB1, TRUB2 Other modifications (m7G) CMTR1, CMTR2, RNGTT, RNMT |

康成使用Arraystar的芯片为客户提供服务,以下为相关资料:
产品说明书: Manual_NuRNA™_Human_Epitranscriptomics_PCR_Array_v1.02
Manual_NuRNA™_Human_Epitranscriptomics_PCR_Array_v1.02
分析工具: Analysis Tool for NuRNA™ Human Epitranscriptomics PCR Array_v1.0
Analysis Tool for NuRNA™ Human Epitranscriptomics PCR Array_v1.0

